目次
MolMole: Molecule Mining from Scientific Literature
この論文は、科学文献から分子構造や反応データを自動的に抽出するためのビジョンベースの深層学習フレームワーク「MolMole」を提案しています。
MolMoleは、分子検出から光学化学構造認識までを一つの深層学習パイプラインで統合し、化学データの自動抽出を効率的に実現する新しいフレームワークです。
論文:https://arxiv.org/abs/2505.03777
リポジトリ:https://lgai-ddu.github.io/molmole/

以下は、弊社AI開発ツール「IXV」を用いてこの論文を要約したものです。見出しや章立てが元論文とは異なる場合があります。
概要
科学文書からの分子構造および反応データの抽出は、多様で非構造的な化学フォーマットや複雑な文書レイアウトのために困難です。これに対処するために、MolMoleというビジョンベースの深層学習フレームワークを紹介します。このフレームワークは、分子検出、反応図の解析、光学化学構造認識(OCSR)を単一のパイプラインに統合し、ページレベルの文書から化学データの自動抽出を実現します。標準的なページレベルのベンチマークと評価指標が欠如していることを認識し、分子のバウンディングボックス、反応ラベル、およびMOLファイルで注釈を付けた550ページのテストセットと新しい評価指標を提示します。
実験結果は、MolMoleが当社のベンチマークおよび公的データセットの両方で既存のツールキットを上回ることを示しています。このベンチマークテストセットは公開され、MolMoleツールキットはLG AI Researchのウェブサイトでインタラクティブデモを通じて近日中にアクセス可能となる予定です。
1. 序論
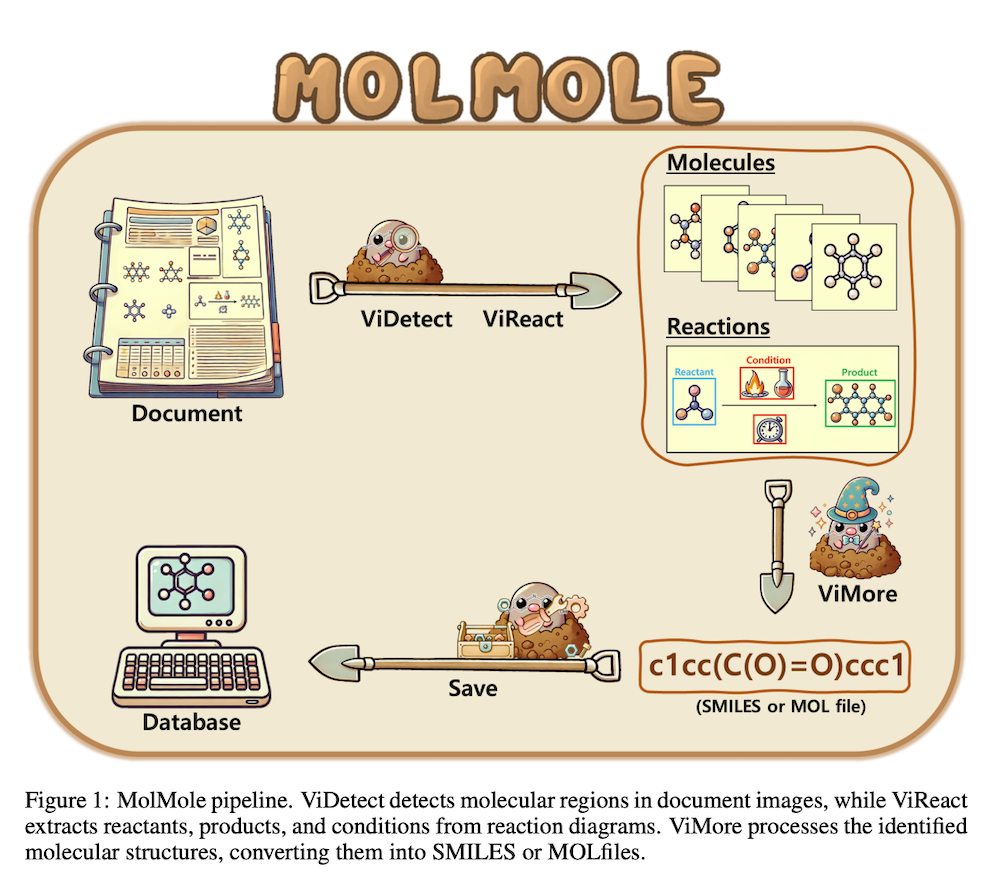
科学出版物の急増により膨大な分子構造・反応データが蓄積されていますが、多くは非構造化形式のままです。既存のフレームワークであるDECIMERは反応ダイアグラム処理ができず、OpenChemIEは外部レイアウトパーサーに依存するという限界があります。本研究では、ページレベルの分子情報抽出のためのディープラーニングツールキット「MolMole」を紹介します。MolMoleはレイアウトパーサー不要で、分子検出(ViDetect)、反応ダイアグラム解析(ViReact)、OCSR(ViMore)を統一ワークフローに統合しています。
2. MolMoleパイプライン
MolMoleはPDFをPNG画像に変換し、ViDetectとViReactで並行処理します。ViDetectは分子構造のバウンディングボックスを検出し、ViReactは反応ダイアグラムを解析して主要コンポーネントを抽出します。分子領域が特定されると、ViMoreが分子画像をMOLfilesやSMILESなどの形式に変換します。最終データはJSONやExcelなど様々な形式で保存できます。
2.1 分子検出のためのViDetect
ViDetectはDINOから派生したオブジェクト検出モデルで、文書画像内の分子構造のバウンディングボックスを予測します。プライベートデータセットでエンドツーエンドでトレーニングされ、ポスト処理で重複する提案を除去します。大規模データ処理のため、DECIMERの計算コストが高いセグメンテーション方式やOpenChemIEの自己回帰アプローチとは異なり、ViDetectはDETRベースのアーキテクチャを採用し、速度と精度のバランスを取りながら効率的な処理を実現します。
2.2 反応ダイアグラム解析のためのViReact
ViReactはページレベルの文書画像から直接反応情報を抽出する深層学習モデルです。RxnScribeアーキテクチャに従い、エンコーダが入力画像を隠れ表現に抽象化し、デコーダが自己回帰的に構造化された反応シーケンスを生成します。既存モデルは切り抜かれた反応ダイアグラムでトレーニングされ、レイアウトパーサーによる前処理が必要ですが、ViReactは前処理不要でページ全体の入力に直接対応します。このアプローチをサポートするために詳細なアノテーションを持つカスタムデータセットを開発しました。
2.3 光学化学構造認識のためのViMore
ViMoreは分子画像を機械可読形式に変換するOCSRモデルです。原子領域を検出し、原子記号を認識し、結合タイプを予測して構造化された分子表現にまとめます。生成モデルと異なり検出ベースのアプローチを採用し、幻覚エラーを避け、解釈可能性を向上させ、レイアウトを考慮したMOLfile生成を可能にします。SMILESの制約を超えて拡張可能で、ポリマー構造のブラケット表記や波線結合を認識できます。また、予測結果に低・中・高の信頼度レベルを付与し、ユーザーが出力の信頼性を評価できるようにしています。
3. 性能
3.1 ベンチマーク
ページレベル抽出評価の課題はエンドツーエンドのベンチマークデータセットの欠如です。OCSRベンチマークは画像から分子への変換のみに焦点を当て、ページレベルで重要な分子検出を評価していません。そこで科学記事と特許から550ページのカスタムデータセットを構築しました。各ページには分子のバウンディングボックス、反応ダイアグラムコンポーネント、MOLfile形式での分子表現の完全なアノテーションがあり、パイプライン全体のエンドツーエンド評価を可能にしています。
3.2 評価
MolMoleはDECIMER 2.0とOpenChemIEと比較評価しました。具体的には、ViDetectはDECIMER SegmentationとMolDetectと、ViMoreはDECIMER Image TransformerとMolScribeと、ViReactはRxnScribeとReactionDataExtractor 2.0と比較されました。
3.2.1 ページレベルの分子検出と認識
評価は(1)分子検出性能、(2)GTバウンディングボックスを使用した分子変換性能、(3)分子検出と変換の組み合わせ性能の3つを行いました。分子検出はAP、AR、F1スコアで評価し、ViDetectはすべてのメトリクスとデータセットで一貫してベースラインモデルを上回りました。分子変換ではViMoreが両方のベンチマークで最高性能を達成し、全パイプラインの評価でもViDetect+ViMoreの組み合わせが最高の精度、再現率、F1スコアを達成しました。
3.2.2 ページレベルの反応ダイアグラム解析
RxnScribeで提案されたハードマッチとソフトマッチの評価メトリクスを採用し、バウンディングボックスのオーバーラップでグラウンドトゥルース反応と比較しました。ViReactはすべてのベースラインモデルをすべてのメトリクスと評価設定で上回り、特許テストセットではソフトF1スコア0.980、ハードF1スコア0.925を達成しました。記事テストセットでも同様の傾向が見られました。
3.2.3 OCSR公開ベンチマーク評価
公開OCSRベンチマークを使用してViMoreを評価し、DECIMER Image Transformer、MolScribe、MolGrapherと比較しました。USPTO、UOB、CLEF、JPOの4つのベンチマークデータセットで実験を行い、ViMoreは4つのうち3つ(CLEF、JPO、USPTO)で最高精度を達成し、それぞれ85.3%、81.5%、93.8%のInChIマッチング精度を記録しました。
4. 議論
MolMoleの質的強みとして、生成モデルは幻覚や化学パターンへのバイアスで非現実的な構造を予測しやすいのに対し、ViMoreは入力から原子と結合を明示的に検出するため、幻覚やバイアスを軽減できます。また、レイアウト保存MOLファイルの生成、ポリマー構造のブラケット表記や波線の正確な識別、二段組み文書での反応情報の抽出能力など、既存モデルにない優位性を持っています。
5. 結論
本研究では科学文書から直接分子構造と反応データを抽出するMolMoleを紹介し、ページレベルのベンチマークデータセットと評価メトリックを提案しました。MolMoleは既存ツールを上回る性能を示し、解釈可能性の向上、レイアウト保存MOLファイル、ポリマーと波線認識の強化、複雑レイアウトでの堅牢な反応解析など重要な利点をもたらします。今後は複雑な分子表現処理の強化とデータセットカバレッジの拡大を目指します。